LSPR生物传感器的计算模拟
了解受体和 TiN 表面之间的分子相互作用,从而实现成功的功能化和随后的生物传感,是计算建模最重要的属性。 在科竟达生物技术有限公司,我们在计算资源上投入了大量资金。 我们的集群上安装了三个开源软件包,即(1)用于基于量子力学的密度泛函理论(DFT)计算的Quantum Espresso,(2)用于基于经典力场的分子动力学的GROMACS,(3)时域有限差分的MEEP对纳米颗粒在等离子体共振下与光波相互作用的电磁建模。 通过这三个软件包,我们可以探索 TiN 激元生物传感器的生物化学反应,从电子层面到分子水平,最后到纳米尺度。
让我们从一个例子开始,即 TiN(200) 表面上寡核苷酸的功能化。 这对于我们用于分子诊断的 LSPR 生物传感器尤其重要。 我们从四种基本核苷酸开始,即腺嘌呤 (A)、高嘌呤 (G)、胞嘧啶 (C) 和尿嘧啶 (U)。 把其中的单个核苷酸放置在足够靠近 TiN(200) 表面的位置,但相对于糖环命名法摆设于不同的方向,即 1' 至 5'。 1’端装饰有 AGCU 之一的单个碱基,负责通过氢键形成 DNA/RNA 双螺旋。 5’端是 PO4 官能团,它负责 DNA/RNA 主链的延伸,从而形成基因序列。 DFT 计算中使用标准赝势库。 该模型包括具有足够多个 TiN 基本单元的 TiN(200) 平板、具有选定方向的寡核苷酸以及平板上方的真空层,以避免相邻镜像之间的相互作用。 该模型完全松弛至基态,系统的最终能量以电子伏特 eV 为单位。 各取向的吸附能按下式计算:
Eads = Eslab+nt - Eslab - Ent
其中,Eslab+nt 是 TiN 板表面吸附有核苷酸的模型的最终能量,Eslab 是仅具有 TiN 平板表面的模型的最终能量,Ent 是仅具有核苷酸的模型的最终能量。 需要指出的是,糖环中从 1' 到 5' 至少有 5 个方向,因此每个核苷酸至少有 5 次计算。 通过吸附能的比较,我们可以估计TiN表面的优先吸附方向。 下表 1 总结了吸附能最引人注目的发现。
|
(eV) |
A |
G |
C |
U |
|
1’ |
-0.54 |
-0.55 |
-0.51 |
-0.83 |
|
5’ |
-1.79 |
-2.34 |
-1.75 |
-2.02 |
表 1、AGCU 在 1' 和 5' 取向 TiN(200) 表面上的吸附能
因此很明显,5'端的吸附能比1'端的吸附能更负。 因此,带有 PO4 的 5' 端是 AGCU 锚定在 TiN(200) 表面上的首选方向。 这实际上有利于 DNA/RNA 传感场景,因为互补序列总是通过氢键通过 1' 碱基杂交。 由于功能化结合优选通过 5' 端,因此它不会与杂交位点竞争结合。 因此,AGCU 核苷酸可以直接用于功能化 TiN 表面,用于 LSPR 生物传感。 这是对现有金基 SPR 装置的改进,因为它即简单又有效。 操作员不再需要使用不稳定的硫醇化学品激活传感表面,从而节省时间和成本。 1'碱基和5'端TiN(200)上AGCU的优化结构分别如图1和图2所示。
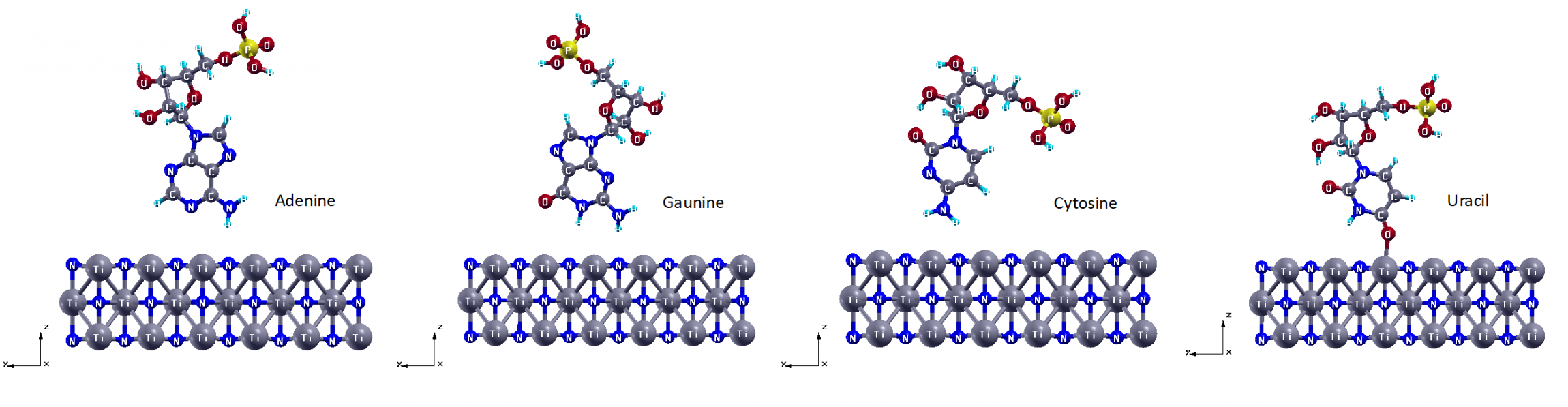 图 1. 通过1' 碱基吸附在TiN(200) 上的核苷酸 AGCU优化结构
图 1. 通过1' 碱基吸附在TiN(200) 上的核苷酸 AGCU优化结构
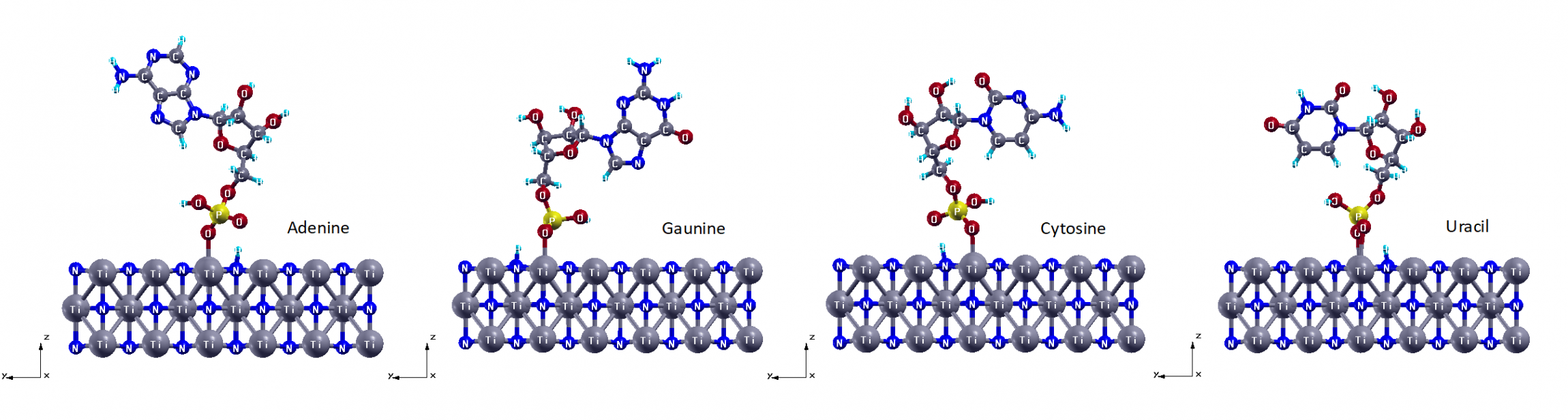
图 2. 通过5' 端吸附在TiN(200) 上的核苷酸 AGCU优化结构
计算模型的另一个强大方面是通过动态对接探索特定蛋白质靶标上的潜在受体候选者。 例如,100 Da 的合成小分子受体在捕获目标蛋白抗原方面比 100 KDa 的抗体更有效。 这是因为大抗体容易受到非特异性结合干扰并给出假阳性结果。 对接过程本质上是通过分子动力学进行的,受体一开始放置在哪里并不重要。 当计算开始时,受体围绕蛋白质靶标随机移动,寻找对接位置。 一旦找到对接位置,受体就会重新调整其位置以适合对接口袋。 之后,只要系统温度保持大致相同,它就会留在口袋中。 为了说明分子动力学的动态对接过程,我们使用 GROMACS 开源包来研究 Kelch样ECH关联蛋白-1 (KEAP-1) 和一种来自草药提取物的名为 LgN 的小分子之间的相互作用。 LgN 与 KEAP1 目标的均方根距离 (RMSD) 计算为 200 纳秒,并绘制在图 3 中。该数字是 MD 模拟运行时 LgN 位置的指示标。 在前 25 纳秒内,LgN 倾向于随机移动以搜索结合靶点。 从 25 到 75 纳秒,RMSD 变化相对较少,因为 LgN 附着在距离初始位置相对较远的位置。 然而,这一个靶点并不稳定。 因此,LgN 与靶点分离并在大约 85 纳秒时重新开始搜索。 从 85 到 105 纳秒,LgN 克服了位置障碍,最终到达了最佳对接口袋靶点。 125 纳秒后,LgN 随着时间的推移保持在相同的对接靶点。 因此,我们确信 LgN 是 KEAP-1 靶点小分子受体的潜在候选者。 RMSD 阶段 A、B 和 C 的快照分别如图 4、5 和 6 所示。 A、B、C阶段相应的动画电影也可以在此页面找到。
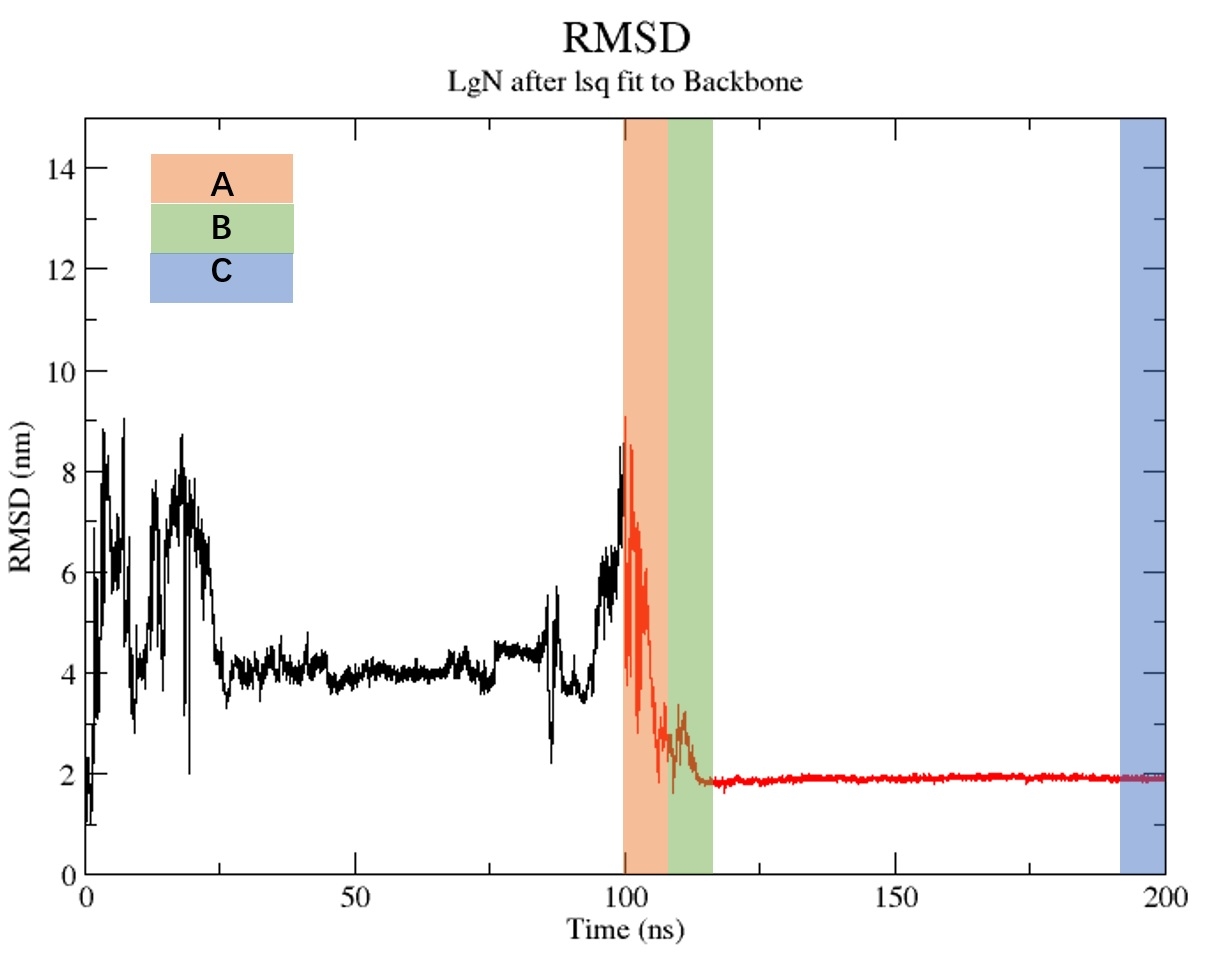
图 3. LgN 的RMSD最小二乘拟合至KEAP-1主干网

图 4. A阶段LgN和KEAP-1 的俯视图。

图 5. B阶段LgN和KEAP-1的俯视图。

图 6. C阶段LgN和KEAP-1的俯视图
最后,开源有限差分时域 (FDTD) 包 MEEP 有助于计算自由空间光波与 TiNC 相互作用并引发等离子体共振时的电磁场。 众所周知,等离子体共振会在纳米结构附近产生增强的电磁场,但很少研究波能的远场重新分布。 然而,远场强度模式在局域表面等离子体共振生物传感器的发展中起着至关重要的作用。 此外,在基板的 3D 打印制造中,我们通过 LED 光泵浦将光激发染料分子作为偶极源引入基板。 这是为了使热等离子加热能够展开 DNA/RNA 序列,以便在 TiNC 表面上直接杂交。 这样做,我们可以使用我们的 LSPR 生物传感器和热等离子技术来检测 DNA/RNA 序列的不同片段。 计算模型包括一个长度为45nm的TiN纳米立方体,它部分嵌入PMMA基板中,如图7所示。通过3D打印,我们可以调整与PMMA单体预混合的染料分子的浓度,因此这里我们修改了 PMMA 基板中的激发偶极子数量从 1 到 10,000。 入射波长设置为 638nm,并在 XZ 平面内偏振并朝 Z 方向传播。 电磁场平面监视器放置在XZ和YZ平面的中心以记录光波的幅度和相位。 计算运行足够长的持续时间,直到波幅衰减到规定的阈值。 结果如图 8 至 12 所示,其中偶极子源的数量为 1 至 10,000。 很明显,由于入射光波是X偏振的,所以Y方向的电磁场强度(|Ey|2)相当弱。 随着偶极子数量从 1 增加到 10,000,|Ey|2 按比例从 0.06 增加到 200 以上。 随着电磁场的增大,焦耳热也会增加。 这肯定了我们的 TiN 纳米立方体是一种适合热等离子应用的优异纳米结构。

图 7. 部分嵌入 PMMA 基板中的 TiN 纳米立方体 3D 模型
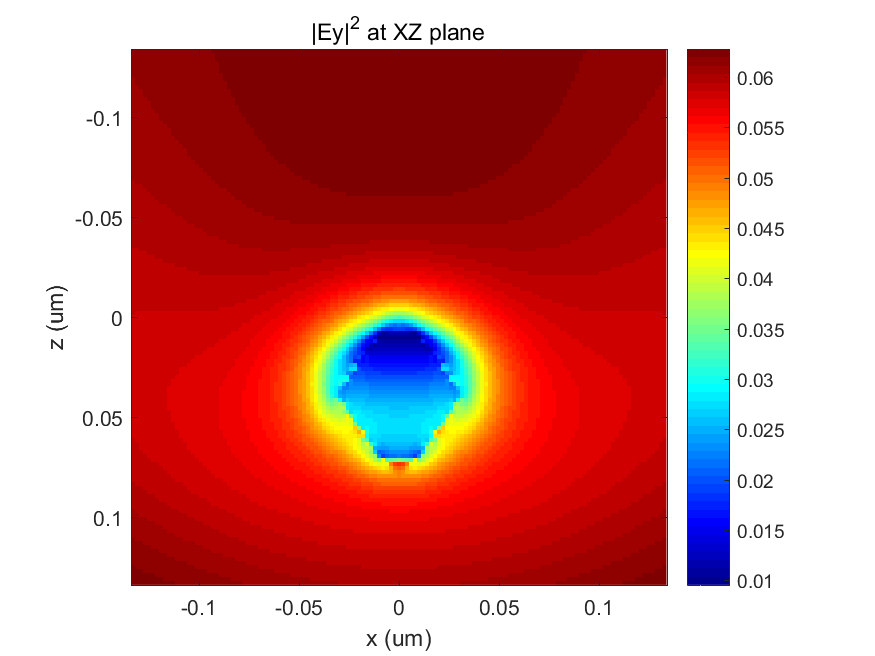
图 8. 使用 1 个偶极源时 TiN 纳米立方体在 XZ 平面上的电磁场强度|Ey|2
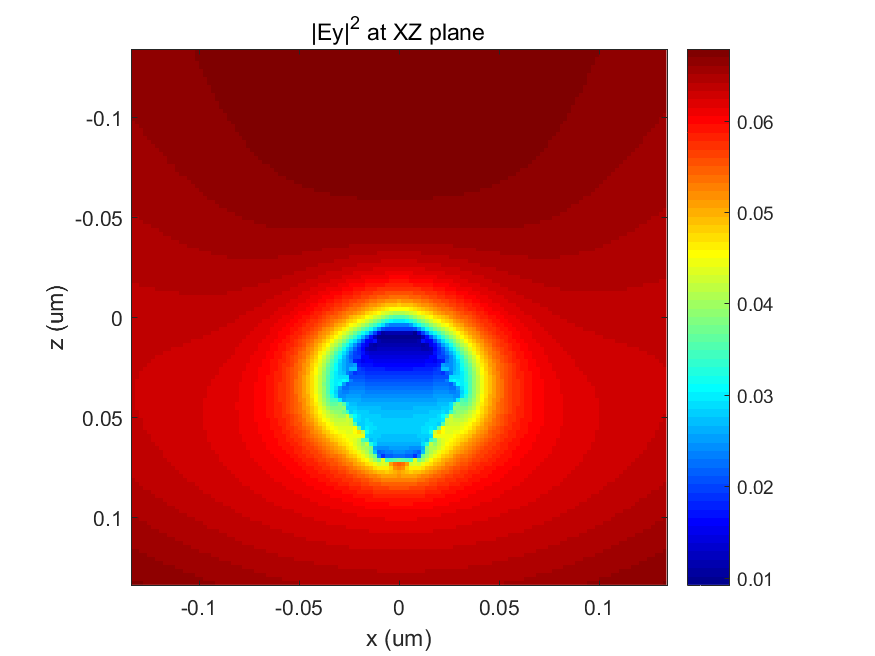
图 9. 使用 10 个偶极源时 TiN 纳米立方体在 XZ 平面上的电磁场强度|Ey|2
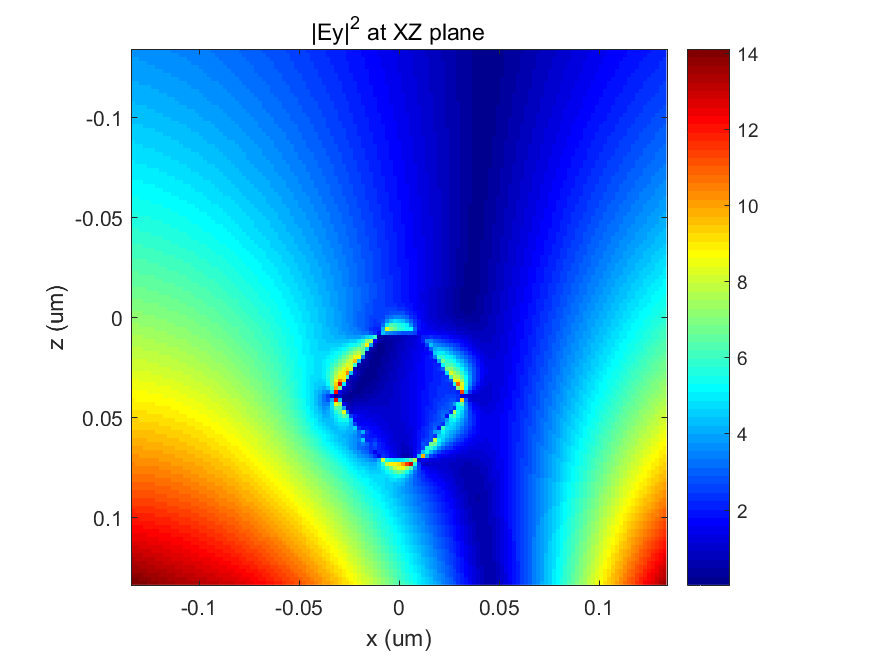
图 10. 使用 100 个偶极源时 TiN 纳米立方体在 XZ 平面上的电磁场强度|Ey|2
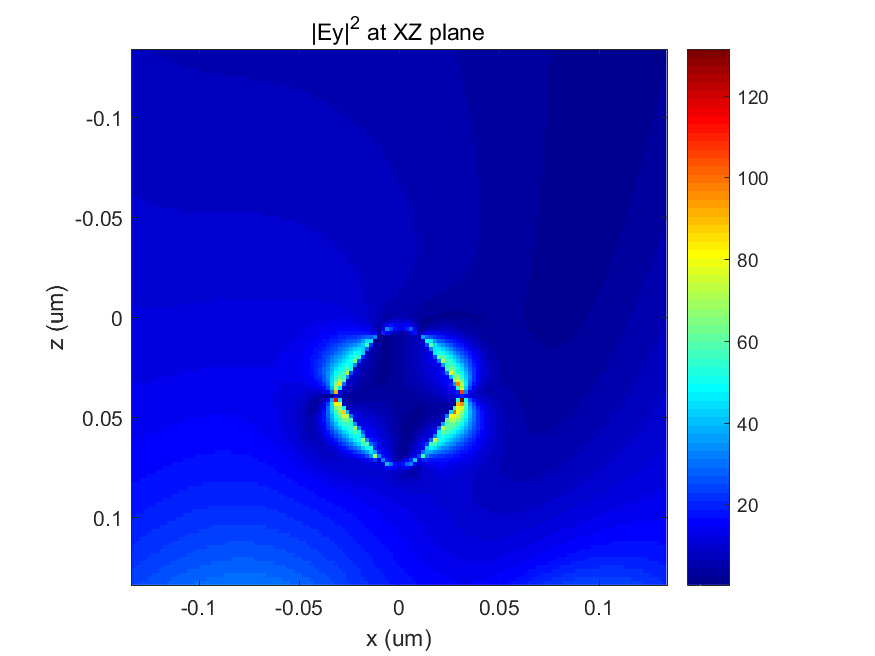
图 11. 使用 1000 个偶极源时 TiN 纳米立方体在 XZ 平面上的电磁场强度|Ey|2
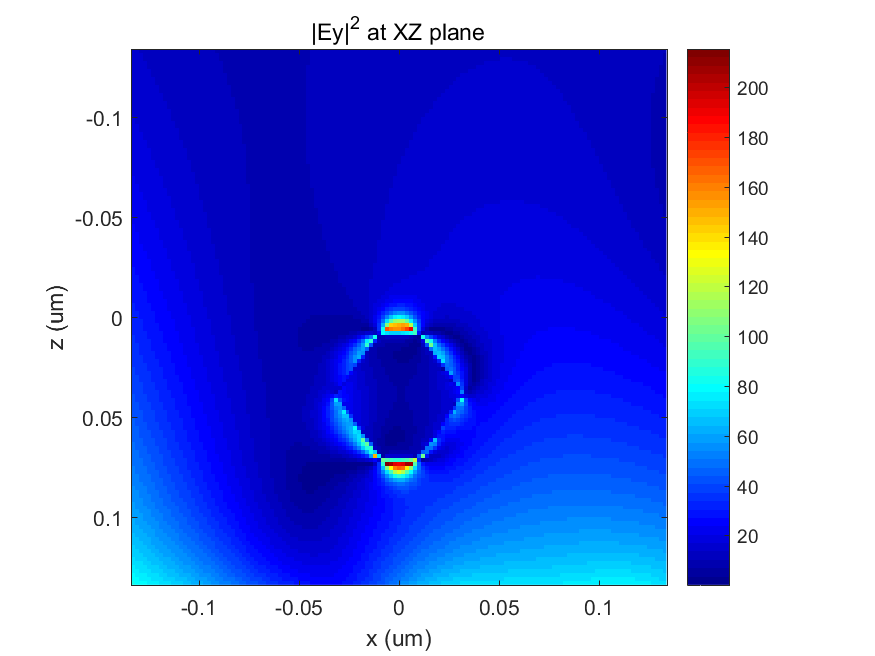
图 12. 使用 10000 个偶极源时 TiN 纳米立方体在 XZ 平面上的电磁场强度|Ey|2
参考文献
[1] Quantum Espresso, https://www.quantum-espresso.org/
[2] GROMACS, https://www.gromacs.org/
[3] MEEP, https://meep.readthedocs.io/en/latest/

