现有的定量逆转录聚合酶链反应(qRT-PCR)核酸检测被认为是检测新型冠状病毒(SAR-CoV-2)的黄金法则。 然而,qRT-PCR在病毒基因组检测方面的局限性也很明显。
- 第一,SAR-CoV-2 基因组由 30,000 个核苷酸组成,此后几乎每个部分都在不断突变。 相反,qRT-PCR探针的长度只有约20个核苷酸,因此很可能会错过发生突变的互补靶标。
- 其次,qRT-PCR 需要聚合酶通过正向和反向引物构建和扩增目标序列。 如果这些病毒片段也发生突变,引物也有可能错过其目标,从而导致检测失败。
- 第三,聚合酶的构建-简并-扩增过程并非没有错误。 因此,它会给扩增产物引入复制错误并产生错误的结果。
- 最后,由于荧光颜色串扰,在单个实验中通过 qRT-PCR 可以检测到的病毒片段的数量相当有限。 因此,迫切需要替代的多重方案来进行高通量多病毒片段检测。
很遗憾是,现有的多重生物传感系统,例如下一代测序(NGS)或单分子阵列(SIMOA)对于临床应用来说非常耗时。 然而,这些系统远未满足四个重要特征,即多处理、快速、精确和成本效益(MQPC,多快好省)。
相反,基因编辑工具在生物传感领域变得越来越流行。 麻省理工学院和哈佛大学的研究人员建立了一种灵敏度达到埃(单拷贝)水平、特异性达到单碱基水平的核酸检测技术,即基于 CRISPR-Cas13a被称为基因剪刀,用于高灵敏度检测SARS-CoV-2病毒的痕量核酸[1, 2]。 在此,我们采用 CRISPR-Cas13 基因编辑技术和相应的 crRNA 组合,将 SARS-CoV-2 病毒刺突基因组分割成片段。 此后,通过热等离子体加热和量子光学测量,通过与互补 RNA 探针直接杂交来检测这些片段。 我们用于 SARS-CoV-2 检测的 QPLoC 生物传感器最具颠覆性的技术突破满足了 多快好省(MQPC) 的要求,其中包括:
- 【多】以无标记方式同时多重检测 100 多个病毒基因组片段,
- 【快】15 分钟内利用热等离激元效应进行快速无标记检测,
- 【好】精确的 SARS-CoV-2 病毒基因组测量,具有阿托摩尔灵敏度,即每毫升 500 个拷贝,
- 【省】通过3D打印氮化钛纳米立方体制成的具有成本效益的生物芯片,以替代黄金材料。
因此,我们与香港城市大学(CityU)一起申请了香港特别行政区政府的创新科技基金(ITF)。 该项目的标题是“用于 SARS-CoV-2 RNA 体外诊断的热等离子芯片实验室微阵列,CRP/039/22”。 为了验证我们的性能,我们使用国际知名制造商的 SARS-CoV-2 qRT-PCR 工具包进行了基准测试,该测试由城大经过培训的人员进行,并在香港科学园(HKSTP)生物医学技术支持中心进行实验。 话不多说,让我们看一下生物传感方案、相应的原始数据以及与 qRT-PCR 的比较。 结果表明,特别是在极低浓度下我们的 QPLoC 生物传感器与 CRISPR-Cas13a 相结合,在检测限和系统线性度方面优于 qRT-PCR。
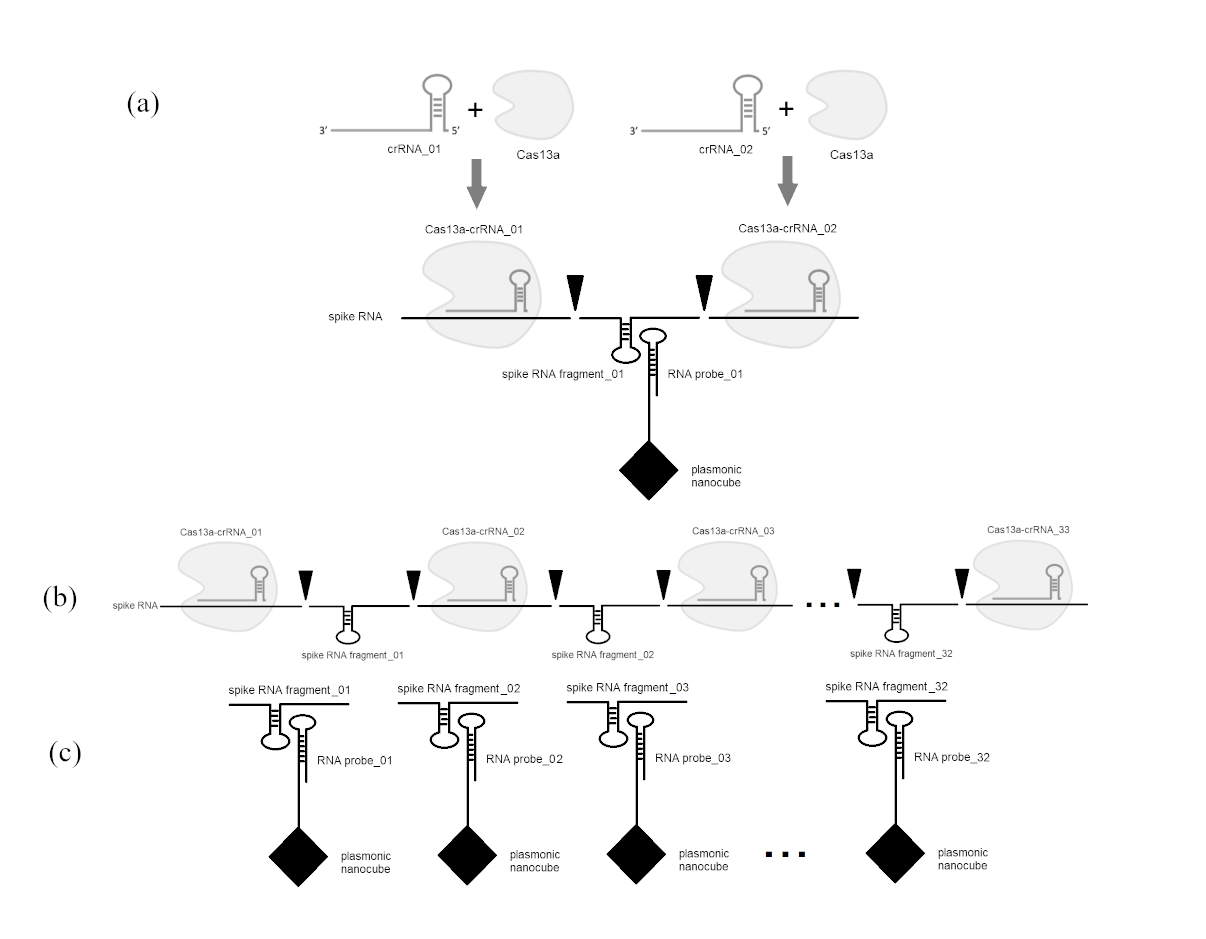
图 1. 使用 TiN 纳米立方体和 CRISPR-Cas13a 对 SARS-CoV-2 刺突 RNA 进行无标记生物传感的示意图,其中 (a) 显示病毒刺突 RNA 被两个剪切 crRNA 和 CRISPR-Cas13a 核酸酶指定的片段分裂。 (b) 显示了 crRNA 和 CRISPR-Cas13a 核酸酶的组合,将长病毒刺突 RNA 分割成多个片段。 (c) 表示通过与 TiN 纳米立方体上功能化的相应 RNA 探针杂交时的热等离激元效应检测到的片段。
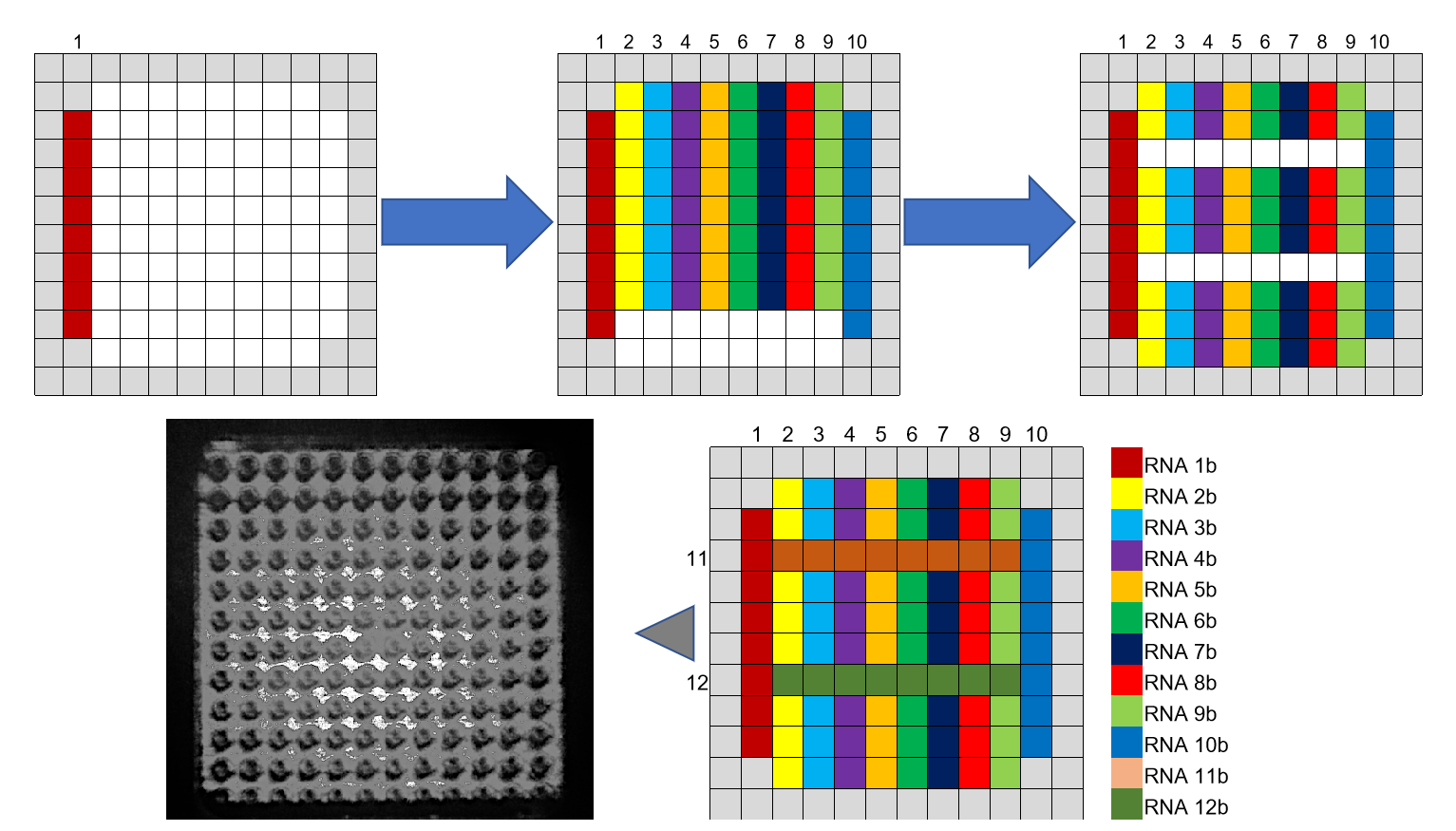 图 2. 生物芯片上不同互补 RNA 探针的相应功能化。
图 2. 生物芯片上不同互补 RNA 探针的相应功能化。
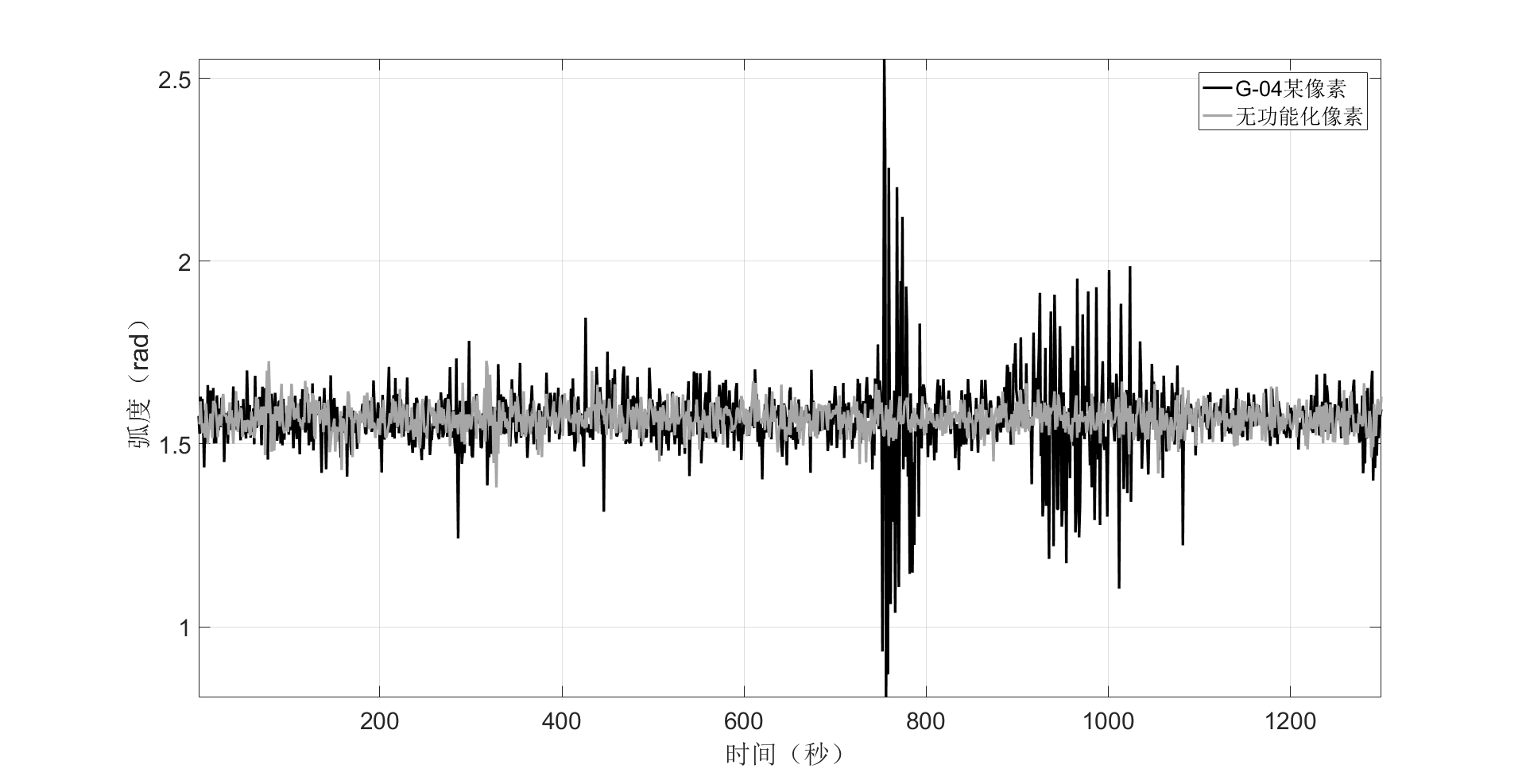
图 3. 有和没有微孔功能化的热等离子杂交的原始数据。 非功能化微孔中的像素没有显示任何响应,而互补功能化微孔中的对应像素在目标-探针杂交中显示出显着的振荡。
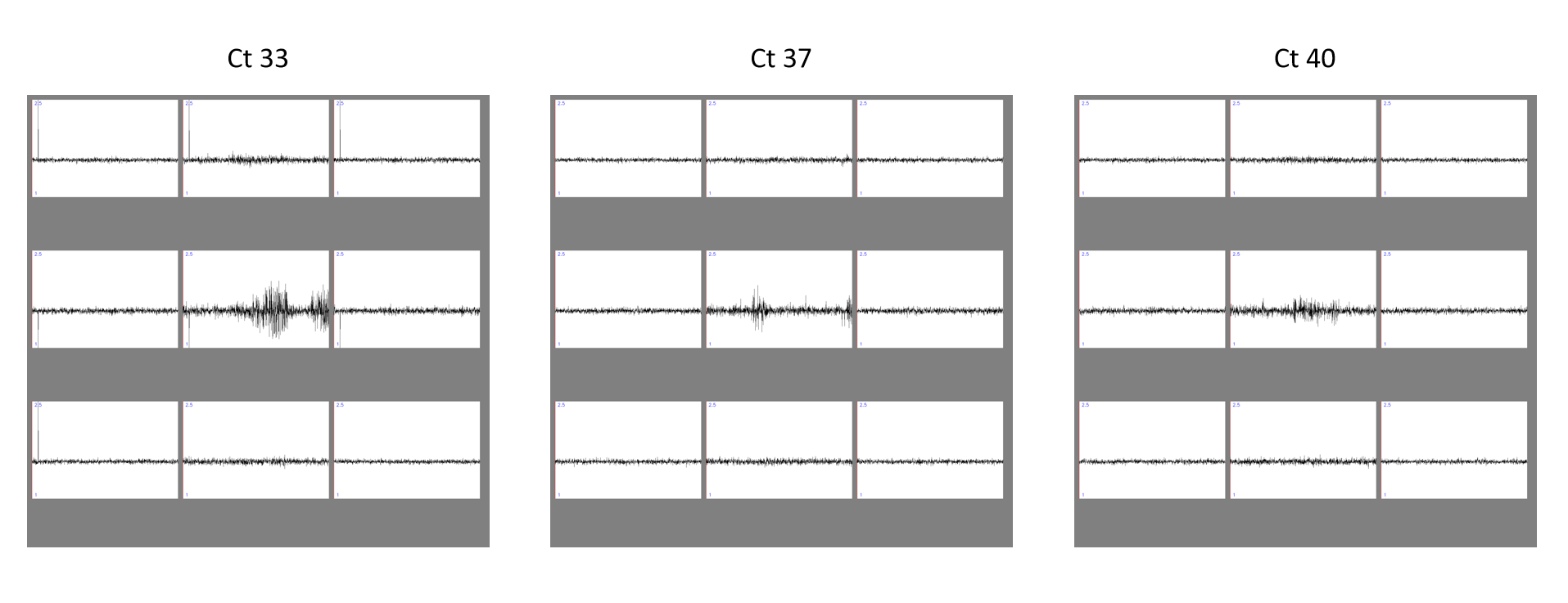
图 4. 不同 Ct 值的阳性对照 SARS-CoV-2 样品溶液中选定像素的原始数据。
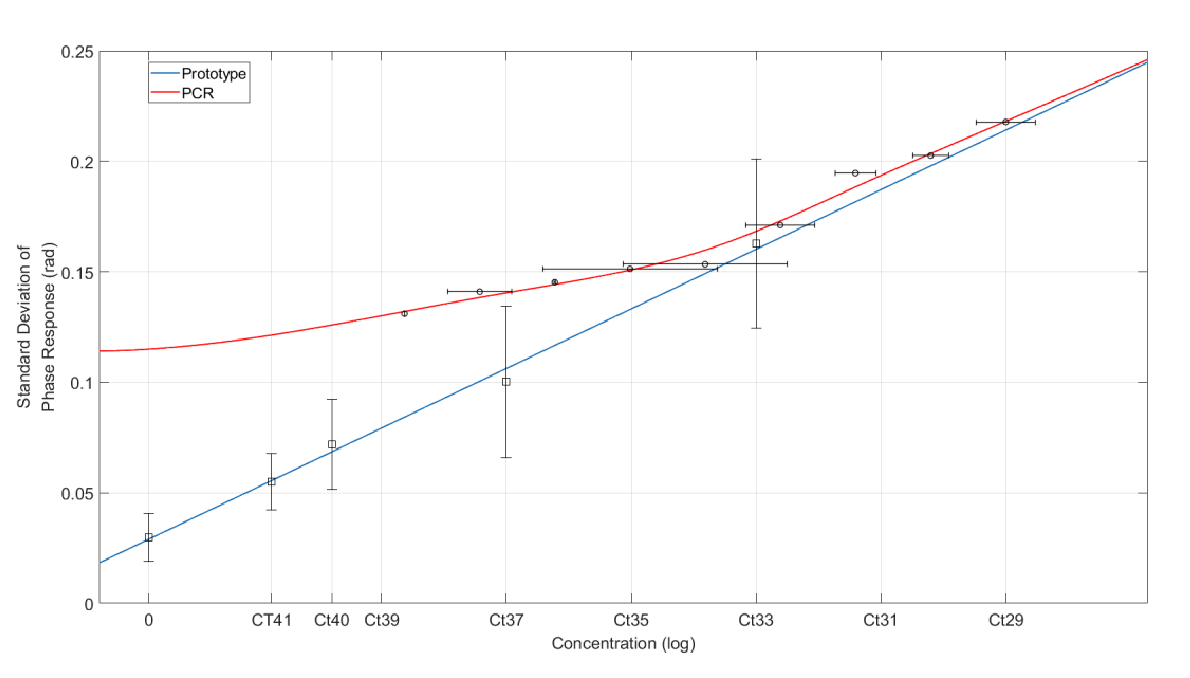
图 5. qRT-PCR 和我们的 QPLoC 生物传感对不同 SARS-CoV-2 浓度的基准比较。 在 QPLoC 生物传感器上观察到低至 Ct 40 的线性,而在 qRT-PCR 上观察到信号饱和。
参考文献
[1] SHERLOCK: nucleic acid detection with CRISPR nucleases
[2] Clinical validation of a Cas13-based assay for the detection of SARS-CoV-2 RNA

